

单细胞转录组数据分析中,细胞类型精准注释一直是瓶颈,传统工具在亚型识别上表现有限。大语言模型(LLM)为这一难题提供了新思路,但缺乏系统基准与标准化流程。为此,张超课题组构建了大规模基准测试,并配套发布开源工具DeepCellSeek。
研究团队对11个主流LLM及三种传统工具进行大规模基准测试,首次系统评估了提示词策略、标记基因筛选方法及模型参数对注释性能的影响,确立了最佳应用策略;随后在34个人和小鼠数据集上进行大规模基准测试。结果显示,大语言模型在细胞类型注释上全面碾压传统工具,尤其在精细亚型识别任务中优势显著;其中 Kimi-k2、GPT-5、Claude-4.1、Grok-4 构成“第一梯队”。团队据此提出“精英集成策略”(Elite Ensemble),整合四个顶级模型并引入细胞本体论校验,实现最优性能与稳定性。
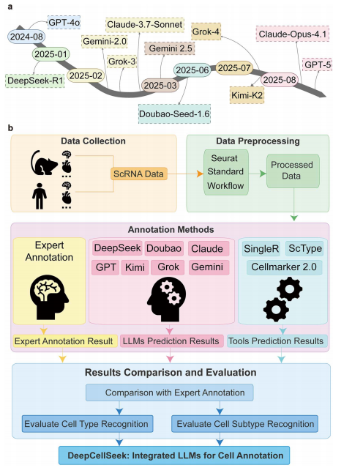
为便于单细胞组学研究者应用,张超课题组同步发布开源R包(https://github.com/ZhangLab-Kiz/DeepCellSeek)与交互式Web平台DeepCellSeek(https://deepcellseek.kiz.ac.cn),提供一站式高精度单细胞注释解决方案。
2025年12月16日,相关成果以 Benchmarking large language models for cell typing in single-cell RNA-seq 为题在线发表于生物信息学著名期刊 Briefings in Bioinformatics。
昆明动物研究所硕士生肖天翔为第一作者,华德志、王亚楠、吕雪梅研究员参与了该项研究,昆明动物研究所张超研究员为论文通讯作者。
该研究工作得到了云南省“兴滇英才支持计划”顶尖团队项目、国家自然科学基金,云南省基础研究计划等项目的资助。
文章链接:
