| 识别非编码区致病性变异研究中取得新进展 |
| 2018-08-29 | 作者: | 来源: 【打印】 |
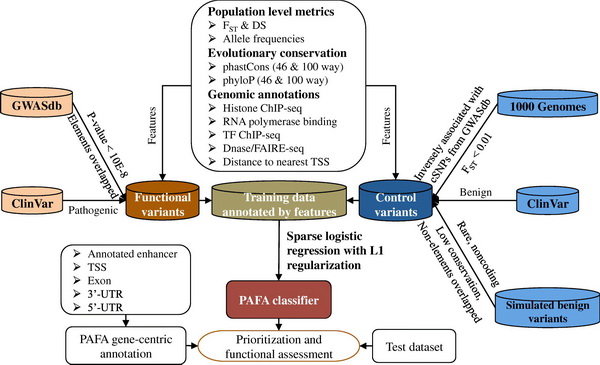
中国科学院北京生命科学研究院、中科院动物进化与遗传前沿交叉卓越创新中心成员赵方庆领导的研究团队首次提出一种全新的基于人群等位基因频率谱的监督集成算法(PAFA),以实现对复杂疾病和性状相关的遗传变异进行打分识别及功能性评估。7月11日,该团队的最新研究成果以Prioritization and functional assessment of noncoding variants associated with complex diseases为题发表在国际学术期刊Genome Medicine上。 PAFA算法引入了丰富的知识库,包括基因组注释、进化保守度指标和人口水平特征。特别是新引入的人群等位基因频率特征值,使得PAFA可以通过计算固定指数和离散度得分,对群体内部及群体间的差异性和多样性进行评估,从而能够在大量的背景变异中有效识别出与复杂疾病有关的功能性变异。此外,PAFA更加合理有效地对训练集进行了数据清洗。它从ClinVar、千人基因组计划和GWASdb等多个权威数据库中获取训练数据,并针对不同来源的变异采取了多重过滤策略。此外,该团队构建了一个友好的在线集成平台。 (访问地址http://159.226.67.237:8080/pafa)
PAFA的方法流程 此平台不仅允许用户利用PAFA对变异进行评估,而且通过整合丰富的功能组学数据,为遗传变异提供全面的功能性注释。通过一系列综合测评分析,PAFA无论对于编码区还是非编码区的变异,都表现出更为出色的功能评估效果。尤其是对于非编码区致病变异的识别方面,比现有方法拥有更高的敏感度和特异度。通过对不同训练特征进行留一法交叉验证,发现群体等位基因频率特征的引入,可以显著提高对非编码区致病变异的识别效率。此外,合理地整合和清洗多种训练集,将会在区分癌症相关的频发变异(recurrent variant)和非频发变异方面取得更好的效果。 |