| 调控网络数学建模揭示干细胞分化关键转录因子 |
| 2019-03-06 | 作者: | 来源: 【打印】 |
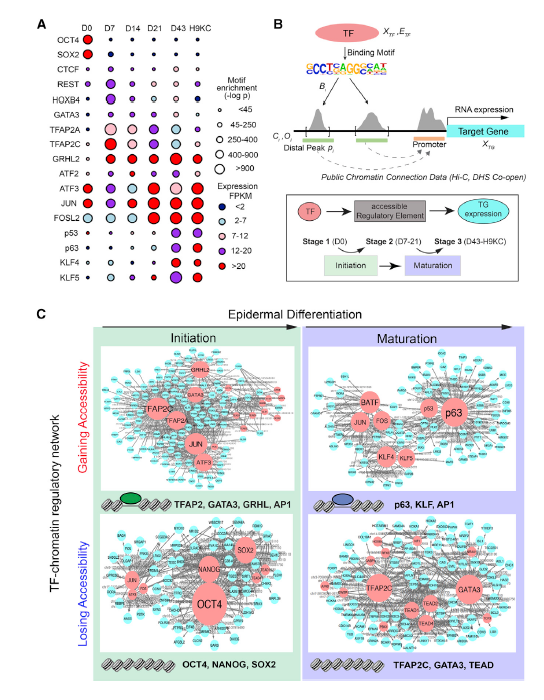
国际权威学术期刊《细胞—干细胞》(Cell Stem Cell)最近在线发表了由中国科学院数学与系统科学研究院和美国斯坦福大学科研人员合作的干细胞分化的基因调控网络建模成果,提出了利用匹配的基因表达和染色质可及性数据刻画转录因子和调控元件结合调控下游基因表达的数学模型,构建了描绘细胞状态转化的染色质调控网络,通过网络分析鉴定出TFAP2C和p63分别为表面外胚层起始和角质形成细胞成熟的关键因子,并揭示了谱系定型过程中TFAP2C-p63对表观遗传转换的反馈调节机制。 干细胞为人类疾病治疗带来了希望,然而如美国国家卫生研究院国立普通医学科学研究所干细胞生物学基金监管人Marion Zatz说:“干细胞研究中面临的最大挑战之一就是如何调控干细胞转变为一种特异的细胞类型。针对这一重要的科学问题,王勇研究员与斯坦福大学统计系Wing Hung Wong教授、医学院皮肤系Anthony Oro教授,Howard Chang 教授,李令杰博士等紧密合作,设计实验获取了从全能性干细胞向表皮角质细胞分化期间的匹配的染色质可及性和基因表达时间序列数据,提出了针对转录因子-可及调控元件-靶基因三元组的网络推断模型,揭示了干细胞分化过程中的两个关键的过渡期:表面外胚层起始阶段和角质形成细胞成熟阶段,并分别将TFAP2C和p63鉴定为谱系起始和成熟的关键转录因子。通过功能实验发现TFAP2C而非p63足以启动表面外胚层分化,TFAP2C启动的祖细胞能够成熟为功能性角质形成细胞。机制上,TFAP2C引发表面外胚层染色质状态变化并诱导p63表达,从而允许成熟因子p63正向自动调节其表达并关闭TFAP2C早期启动的部分原始基因,推动分化过程的完成。该工作提供了一个总体建模框架来推断控制染色质状态转换的调控网络,有望促进未来再生医学的进步。 王勇研究员长期关注对基因表达水平进行精准控制的调控网络的数学建模,之前与合作者发展了转录因子TF,染色质调控因子CR和调控元件RE相互作用网络推断的新方法PECA。PECA重点对转录调控的三个关键环节进行建模:一,基于CR与序列特异性TF 的相互作用推断CR 在RE 上的结合位点;二,基于CR 的结合和RE 的可及性,预测RE 的激活状态;三,基于激活RE 上结合的TF 预测目标基因的表达。PECA应用到"DNA元件百科全书"计划 (Encyclopedia of DNA elements,简称ENCODE)中的多细胞类型的匹配的染色质状态和基因表达数据上,构建了细胞特异的调控网络。此次针对干细胞分化的时间序列数据的建模,发现重要转录因子及其反馈调控机制,是该建模框架的又一成功应用。用数学模型来解读这些重要的数据,将极大地促进我们对后基因组时代基因调控网络的理解。
(左):基于干细胞分化过程的转录因子-可及调控元件-靶基因三元组的网络推断模型;(右):表皮角质细胞分化起始和成熟阶段的调控网络。 这项研究得到中科院B类先导专项、国家自然基金委的经费资助。中科院数学与系统科学研究院的研究生都仁扎那、信晶雪、陈浪参与了数据建模与计算。 |