| 基于单细胞转录组数据构建细胞特异性网络”的研究成果 |
| 2019-03-25 | 作者: | 来源: 【打印】 |
2019年3月13日,国际学术期刊《Nucleic Acids Research》在线发表了中心陈洛南研究组题为“Cell-specific network constructed by single-cell RNA sequencing data”的最新研究成果。该成果首次提出从单细胞转录组数据(scRNA-seq)为每个单细胞构建一个基因关联网络(CSN: Cell-specific network)的理论和方法,使我们第一次能够在单细胞分辨率水平上识别基因之间的相互关联(网络)。利用该方法,可以从网络的角度对scRNA-seq数据进行聚类和拟轨迹分析,为scRNA-seq数据分析开辟了新的途径,并且,该方法还能够发现在网络层面起重要作用但通常被传统的差异表达分析所忽略的“暗”基因,其准确性和鲁棒性在多个scRNA-seq公开数据集中得到验证。 单细胞转录组测序提供了一种高通量方法来测量和比较在单细胞分辨率水平下的基因表达水平,从而揭示了细胞之间的异质性和功能多样性,帮助发现具有独特功能的新细胞类型。而基于单细胞数据中庞大的样本量,理论上我们还可以从这些数据中构建基因关联网络,并在更深层次发现隐含的基因调控关系的变化规律。
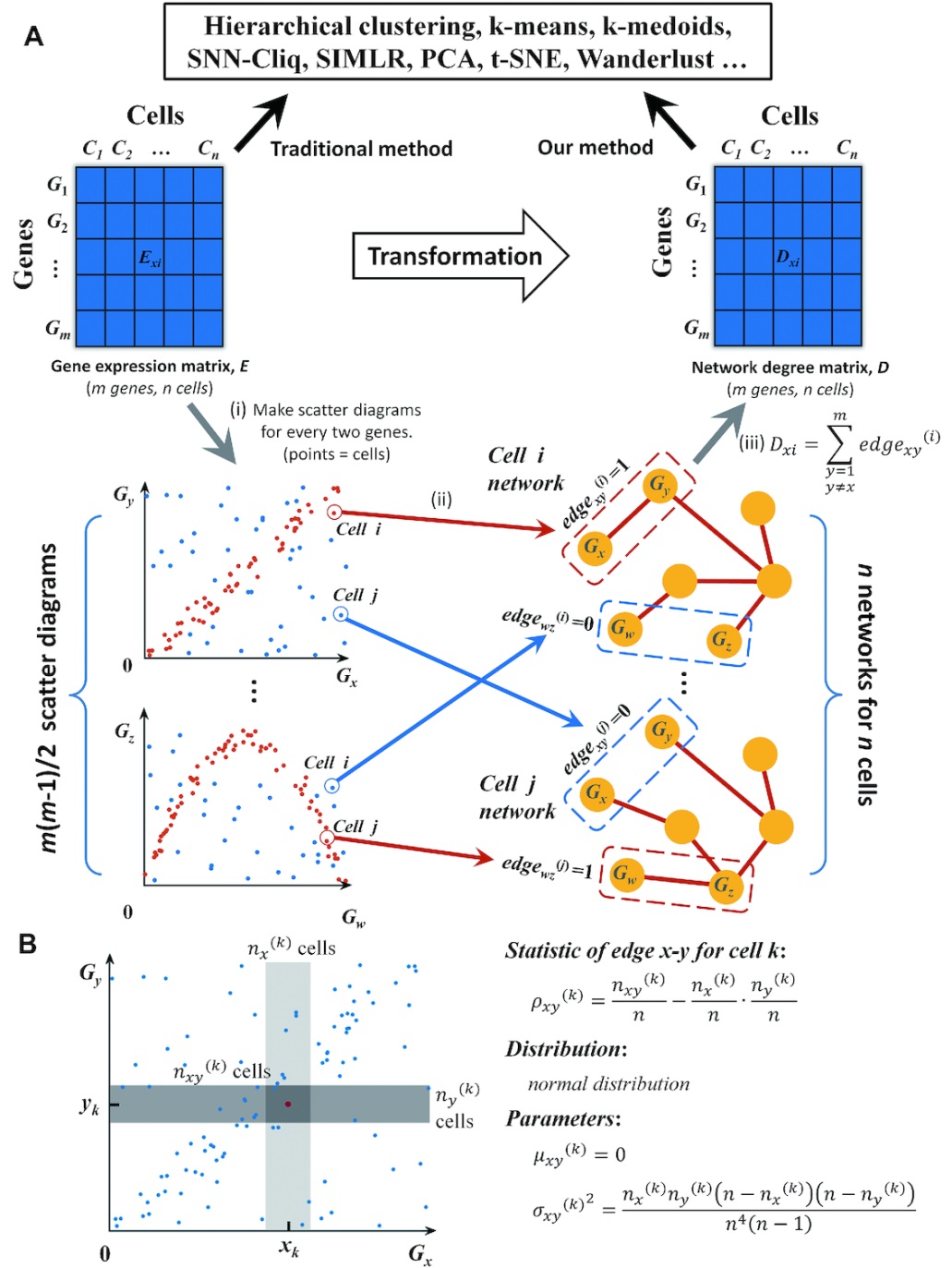
Schematic illustration of CSN and NDM construction and our statistic model. (A) CSN and NDM construction. (i) Make scatter diagrams for every two genes, where each point represents a cell, and x- and y-values are the expression values of the two genes in the ncells. Then mgenes lead to m(m– 1)/2 scatter diagrams. (ii) In the scatter diagram of genes xand y, the plot iwith red color means there is an edge between genes xand yin the cell inetwork based on our statistic model, and if the plot is blue, there is no edge. Then, we can construct ncell-specific networks corresponding to ncells, respectively. (iii) By counting the number of edges connected to each gene in each CSN, we can get the network degree matrix, which is still comprised of mrows and ncolumns, as the same as GEM, and thus it can be analyzed by any existing method. (B) Our statistic model for edge between genes xand y. Near the plot or cell k, make the light and medium grey boxes to represent the neighborhood of xkand ykrespectively. The intersection of two boxes is the dark grey box, which represents the neighborhood of (xk, yk). The number of plots in the light, medium and dark grey boxes is nx(k),ny(k)and nxy(k)respectively. Design the statistic as ρxy(k). If xand yare independent of each other, the statistic follows normal distribution and the mean value and variance can be calculated. If the statistic ρxy(k)is larger than a significant level, label plot kwith red color, which means there is an edge between xand yin cell k; otherwise there is no edge. 为此,我们提出了一种在单细胞水平上构造每个细胞特异网络的新方法,其来源于基于我们关于统计相关性的新理论模型,可以看作是从“不稳定”基因表达数据到“稳定”基因关联数据的转换。计算上,不需要对细胞事先进行聚类或分类,并可以识别基因之间的线性和非线性关联。对多个scRNA-seq数据集的实验均表明,该方法的准确性和鲁棒性优于传统方法,还能发现一些基因在网络水平而非表达水平上存在显著差异,从而在网络层面上提取更丰富的生物系统信息。 生化与细胞所戴昊博士为本文第一作者,该研究得到了中国科学院B类先导、科技部和国家自然科学基金的经费支持。 文章链接 https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkz172/5377474?guestAccessKey=068ea831-989c-42c9-9887-a4783f0616d7 |